La physique hadronique, en tant que sous-domaine de la physique des particules, joue un rôle crucial dans notre compréhension de la matière et des forces fondamentales de l’univers. Les hadrons, tels que les protons et les neutrons, sont des composantes essentielles du noyau atomique. Ils sont composés de quarks, maintenus ensemble par l’interaction forte, l’une des quatre interactions fondamentales de la nature. L’étude de ces particules et de leur comportement dans différents environnements permet de développer des technologies avancées, telles que la protonthérapie, utilisée dans le traitement du cancer [1]. Dans ce court papier, une méthode originale sera proposée pour témoigner du dépôt d’énergie non linéaire des protons qui interagissent avec la matière par interaction électromagnétique.
1. Notion de pouvoir d’arrêt
Les hadrons, et en particulier les protons, ont un comportement tout à fait intéressant concernant leur dépôt d’énergie dans la matière. Ce dernier est caractérisé par le pouvoir d’arrêt, qui décrit la quantité d’énergie déposée dans le milieu par unité de longueur traversée. Il dépend de l’énergie cinétique des protons, de la nature du matériau traversé et il varie de façon non linéaire. En effet, ce dépôt d’énergie lorsqu’il est représenté en fonction de la profondeur dans le milieu, est caractérisé par une courbe qui possède un maximum piqué en fin de parcours, que l’on appelle pic de Bragg.
La Figure 1 illustre le pic de Bragg pour des protons de 3 MeV déposant leur énergie dans de l’eau. Les protons, étant des particules chargées positivement, peuvent être accélérés par une tension électrique. De telles énergies sont obtenues en soumettant les protons à une différence de potentiel de 3 Méga Volts avec l’accélérateur de particules AIFIRA au LP2I à Gradignan [2].
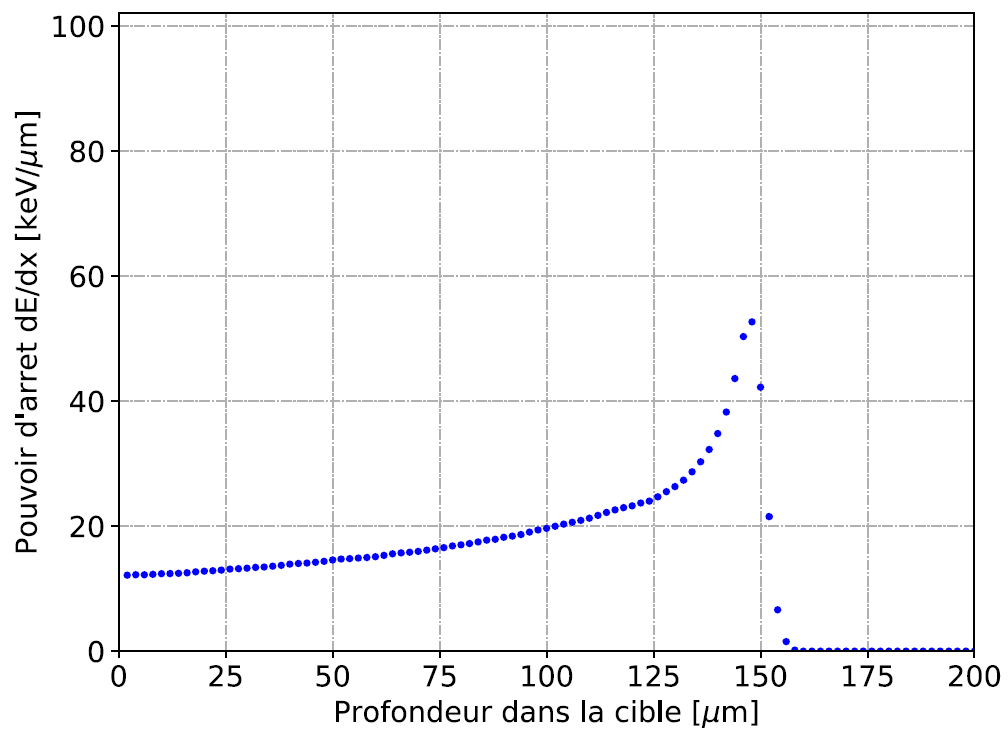
FIGURE 1 – Pic de Bragg - Protons de 3Mev
Il est possible d’observer sur la Figure 1 que lorsque les protons traversent une cible d’eau fine, de l’ordre de 50μm, le pouvoir d’arrêt est presque constant avec une valeur d’environ
dE/dx ≃ 15 keV/μm. En revanche, si la cible est plus épaisse, les protons déposent une quantité croissante d’énergie au fur et à mesure de leur progression, atteignant un maximum à une
profondeur de 150 μm. Après ce point, le pouvoir d’arrêt chute brusquement, signifiant que les protons de 3 MeV ne peuvent en moyenne pas pénétrer au-delà de 150 μm dans l’eau. En effet, au voisinage du pic de Bragg les protons ont des vitesses proches de celles des électrons du milieu qu’ils traversent, ce qui se traduit par un maximum d’interactions électromagnétiques.
Ce comportement des protons est exploité en proton-thérapie. En effet, cette technique permet de cibler précisément des cellules cancéreuses situées à une certaine profondeur, tout en minimisant les dommages aux tissus sains (muscle, tissu, peau) traversés avant d’atteindre la tumeur. Ainsi, le dépôt d’énergie est maximisé sur les cellules cancéreuses, augmentant l’efficacité du traitement tout en réduisant les effets secondaires.
Dans le cadre de mes travaux de thèse, je filme la fragmentation proton-induite de l’ADN en fonction de l’épaisseur d’eau traversée. Je développe par ailleurs un outil de suivi numérique de ces molécules [3], ce qui me permet d’étudier le taux de fragmentation et donc de sonder le pic de Bragg.
2. Fragmentation de l’ADN en solution et pic de Bragg
Le virus bactériophage T4 possède une très longue chaîne d’ADN, de 166 000 paires de bases azotées. Etiré, il mesure 56μm. Il est possible de casser cet ADN avec des protons de haute énergie. De plus, cet ADN est suffisamment gros pour être marqué avec un intercalant fluorescent, le YOYO-1, et observé au microscope par fluorescence. La LED du microscope excite les molécules fluorescentes fixées sur l’ADN à une longueur d’onde de 470 nm, et ces dernières réémettent des photons à 490 nm, ce qui permet de voir l’ADN en solution.
Après avoir déposé cet ADN dans une solution contenant de l’eau pure et un tampon biologique, et s’être assuré que l’intercalant fluorescent de l’ADN et le solvant utilisé ne perturbent pas excessivement la fragmentation, il est possible d’observer comment évolue la probabilité de casser de l’ADN à différentes profondeurs. En effet, plus nous sommes proches de la fenêtre d’entrée des protons, moins ils déposent d’énergie, et donc moins l’ADN est susceptible de se casser. À mesure que l’on observe plus loin dans l’échantillon, les protons déposent de plus en plus d’énergie, augmentant ainsi la probabilité de cassure de l’ADN. Enfin, si nous observons au-delà du pic de Bragg, nous ne devrions plus observer de fragmentation car il n’y a plus de protons.
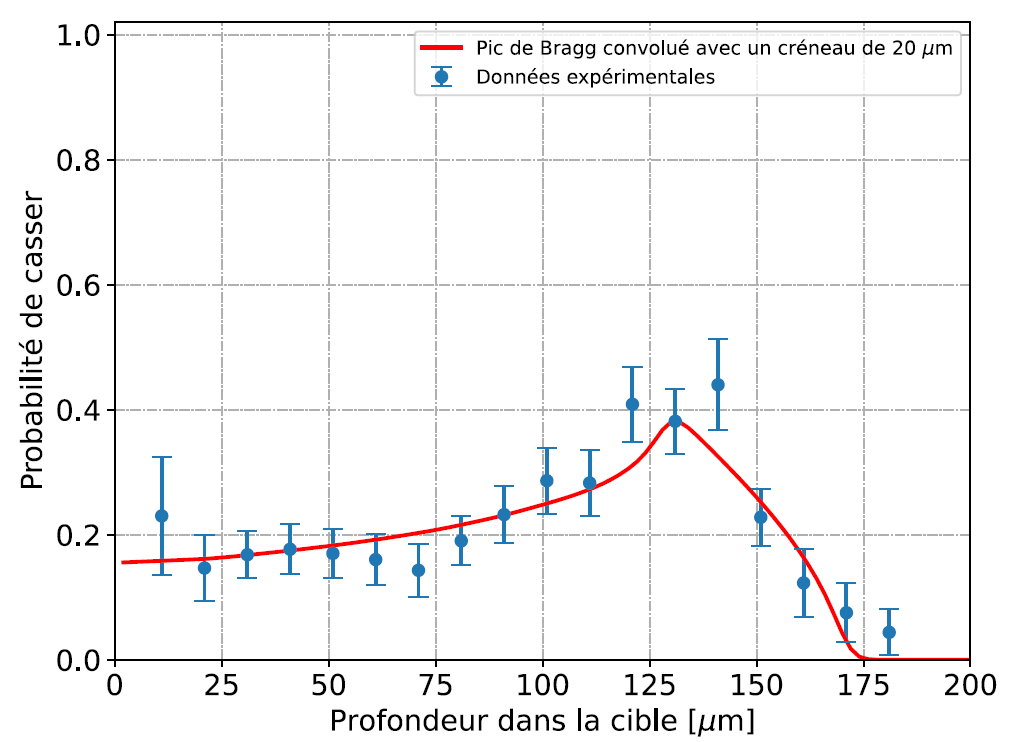
FIGURE 2 – Probabilité de casser une molécule d’ADN en fonction de l’épaisseur en eau
Grâce à un ensemble de codes Python et de machine learning pour l’analyse des vidéos, il est possible de déterminer le nombre de molécules qui se cassent lors de l’irradiation, et donc de calculer la probabilité pour l’ADN de se casser en deux morceaux ou plus, en fonction de la profondeur. La courbe obtenue ressemble beaucoup au pic de Bragg, avec néanmoins un maximum légèrement décalé à de plus faibles profondeurs et un pic moins marqué. Cela s’explique par le fait que le microscope a une profondeur de champ de 10μm, que l’on procède à une moyenne glissante également de 10μm, ce qui introduit une incertitude sur l’épaisseur en eau d’environ 20μm.
En termes plus techniques, les probabilités mesurées sur la Figure 2 sont le résultat d’un produit de convolution entre les probabilités réelles de fragmentation et la réponse au protocole de mesure. A titre de comparaison, la convolution entre le pic de Bragg de la Figure 1 et une fonction porte de largeur 20μm correspond à la courbe rouge de la Figure 2, et elle est en accord avec les données expérimentales. Ces dernières portent donc toutes les informations pour en extraire des taux de fragmentation, après une déconvolution qui reste à réaliser. Ces taux pourront être comparés à ceux donnés par des codes de calcul Monte-Carlo de type Géant4-DNA [4] qui visent à modéliser l’intéraction particules-matière dans les milieux biologiques.
3. Conclusion
Dans ce travail, nous avons illustré une méthode originale pour retrouver le pic de Bragg en utilisant l’irradiation de l’ADN de bactériophage T4 marqué avec un intercalant fluorescent. En observant la fragmentation de l’ADN à différentes profondeurs, nous avons pu obtenir une courbe expérimentale ressemblant au pic de Bragg, malgré certaines limitations liées à la profondeur de champ du microscope.
Cette méthode innovante non seulement confirme les prédictions théoriques du pic de Bragg, mais elle ouvre également de nouvelles perspectives pour étudier les interactions proton-matière à des échelles microscopiques. De plus, l’utilisation de techniques de machine learning pour analyser les résultats démontre le potentiel de l’informatique avancée dans la recherche en physique de la matière molle et en biologie.
En conclusion, la combinaison de la physique des particules/de la matière molle, de la biologie moléculaire et des technologies de l’information constitue un puissant outil pour améliorer notre compréhension des phénomènes complexes et développer des applications thérapeutiques efficaces et précises.
Références
[1] Marco Durante and Jay S Loeffler. Charged particles in radiation oncology. Nature reviews Clinical oncology, 7(1) :37–43, 2010.
[2] P Barberet, J Jouve, S Sorieul, P Alfaurt, and L Mathieu. Aifira : a light ion beam facility for ion beam analysis and irradiation. The European Physical Journal Plus, 136 :1–10, 2021.
[3] John C Crocker and David G Grier. Methods of digital video microscopy for colloidal studies. Journal of colloid and interface science, 179(1) :298–310, 1996.
[4] Sebastien Incerti et al. Geant4-dna example applications for track structure simulations in liquid water : a report from the geant4-dna project. Medical physics, 45(8) :e722–e739, 2018.


